Virologie
MENUPollution chimique et immunité innée antivirale : des liaisons dangereuses ? Volume 22, numéro 1, Janvier-Février 2018
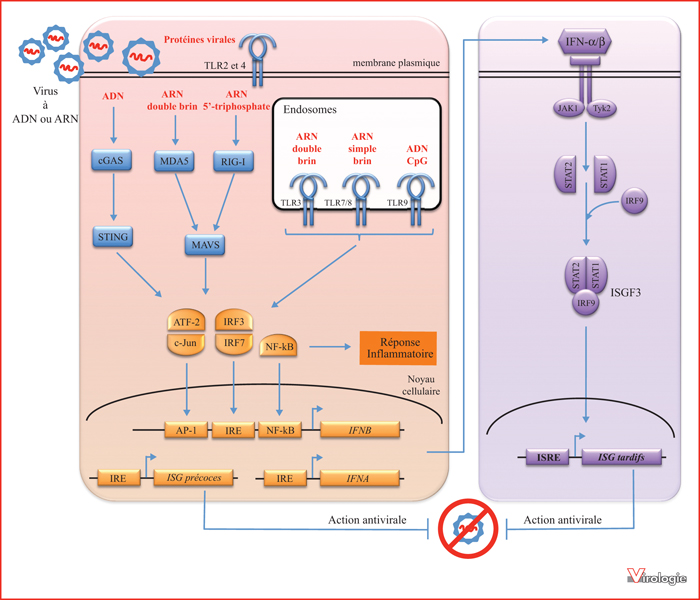
Figure 1
Schéma de la réponse IFN-I. Au contact d’un virus, des récepteurs cytosoliques (cGAS, MDA5, RIG-I) et membranaires (TLR2/3/4/7/8/9) vont être activés par les glycoprotéines d’enveloppe du virus ou les acides nucléiques présents dans la particule virale. Les cascades de signalisation en aval de ces récepteurs convergent vers un ensemble de gènes codant pour des facteurs cellulaires antiviraux, les ISGs précoces, et induisent l’expression des IFN-I. Ces derniers sont secrétés, et stimulent de façon autocrine et paracrine, l’activation des facteurs de transcription STAT1 et 2 qui, en association avec IRF9, contrôlent l’expression d’une seconde vague de gènes antiviraux, les ISGs tardifs. Parallèlement, l’engagement des PRR conduit, via notamment NF-κB et AP-1, à l’expression de facteurs pro-inflammatoires.
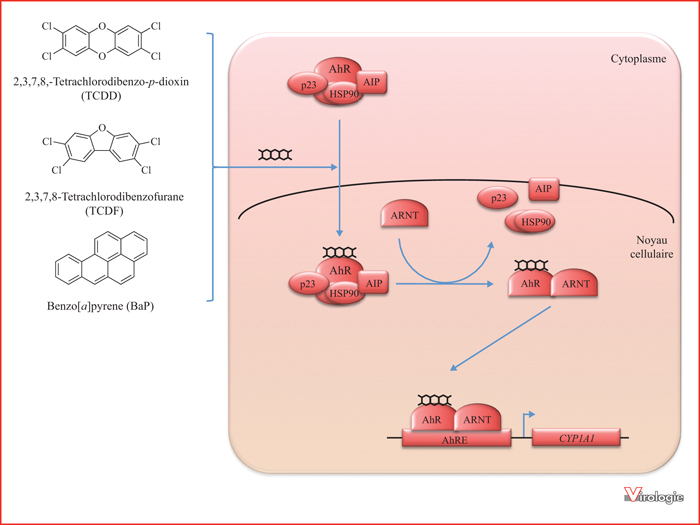
Figure 2
Schéma d’activation d’AhR par certains polluants organiques. En absence de ligand, la protéine AhR est séquestrée dans le cytosol où elle est associée à p23, AIP et aux HSP90. En présence de TCDD, de TCDF ou de BaP, AhR se dissocie des protéines p23, AIP et HSP90 dans le noyau ou le cytoplasme (des doutes subsistent sur ce point) et subit une translocation dans le noyau où il se lie à la protéine ARNT. Le complexe AhR-ARNT se fixe alors sur les éléments de réponse à AhR (AhRE), et induit la transcription des gènes cibles comme CYP1A1.
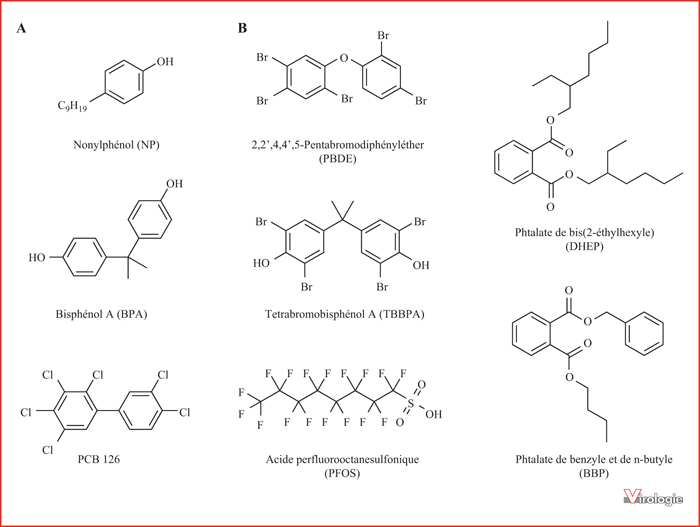
Figure 3
Structures de polluants organiques présents dans les matériaux du quotidien. (A) Le nonylphénol, le bisphénol A et le PCB 126 sont des perturbateurs endocriniens avérés. (B) Structures d’autres molécules polluantes utilisées comme retardateurs de flamme (PBDE, TBBPA), imperméabilisants (PFOS) ou durcisseurs dans les plastiques (phtalates).
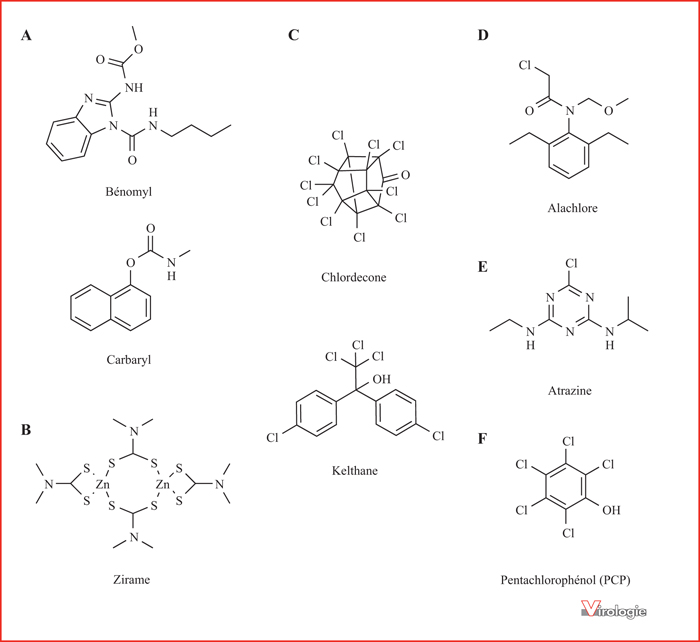
Figure 4
Structures de différents pesticides qui affectent la réponse innée antivirale. (A) Structures du bénomyl (fongicide) et du carbaryl (insecticide) de la famille des carbamates. (B) Structure du zirame, un fongicide de la famille des dithiocarbamates. (C) Structure de deux insecticides organochlorés, le chlordécone et le Kelthane. (D) Structure de l’alachlore, un herbicide de la famille des chloroacétamides. (E) Structure de l’atrazine, un herbicide de la famille des triazines. (F) Structure du pentachlorophénol qui peut être utilisé comme fongicide dans différents matériaux.
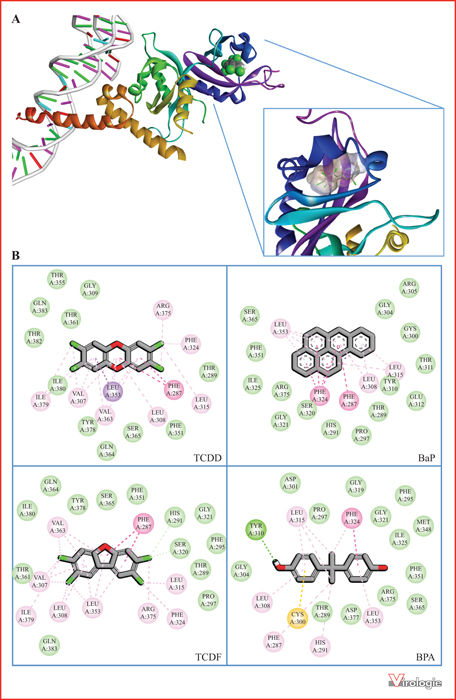
Figure 5
Docking de ligands sur un modèle par homologie d’AhR. (A) La représentation 3D de la protéine AhR a été construite par homologie à partir d’une structure partielle connue d’AhR (pdb code 5NJ8) et de la protéine NPAS3 (pdb 5SY7). Les composés ont été dockés sur la zone d’interaction définie par Goryo et al.[59] à l’aide de cDocker (protéine rigide/ligand flexible). (B) Les résidus impliqués dans l’interaction ont été identifiés et sont illustrés dans les diagrammes d’interaction 2D pour la TCDD, le BaP, le TCDF et le BPA. Ils sont colorés en fonction du type d’interaction effectué avec le ligand : vert pour Van der Waals ; rose pale, rose et violet pour hydrophobes de type alkyl, π-π ou π-σ ; jaune pour sulfur-π.

