Innovations & Thérapeutiques en Oncologie
MENULa transcriptomique spatiale de coupes histologiques : une technique de dernière génération pour le diagnostic médical Volume 10, numéro 2, Mars-Avril 2024
Illustrations
- Mots-clés : transcriptomique spatiale, tissus, cellules, hybridation in situ, séquençage
- DOI : 10.1684/ito.2024.414
- Page(s) : 93-103
- Année de parution : 2024
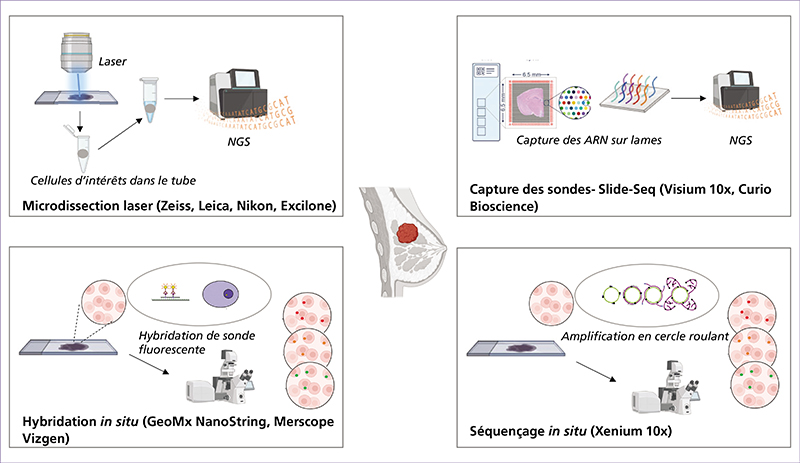
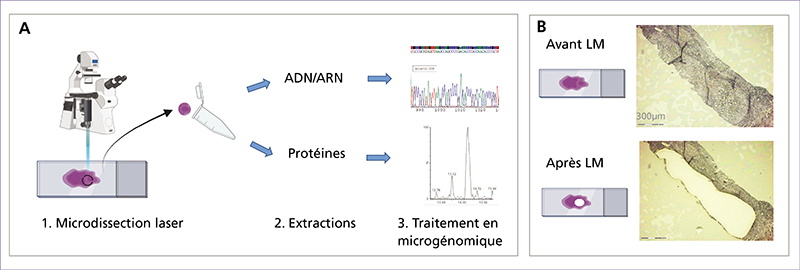
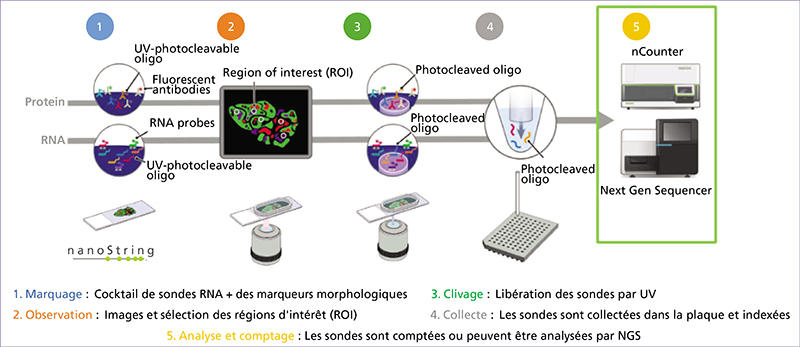
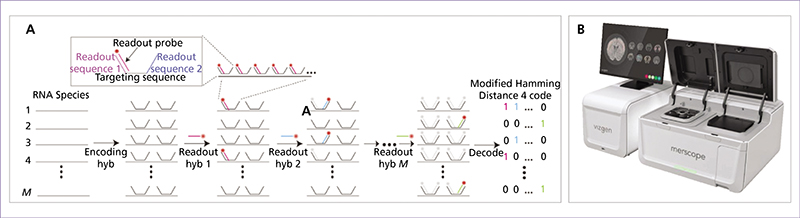
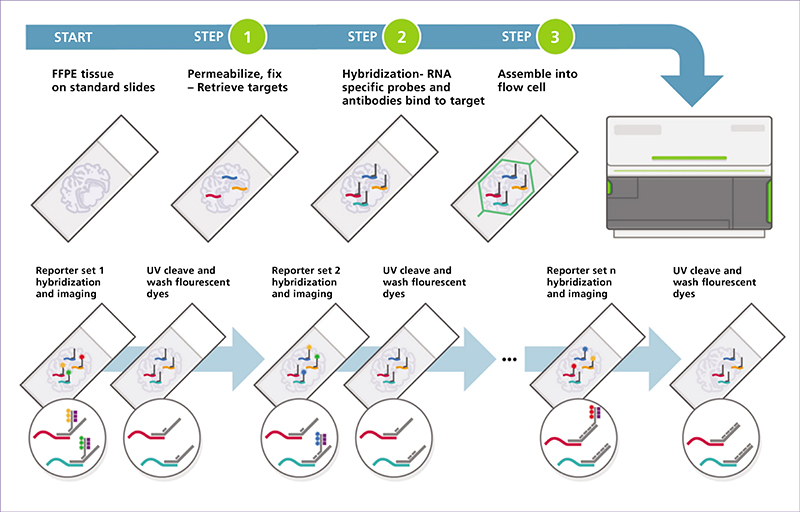
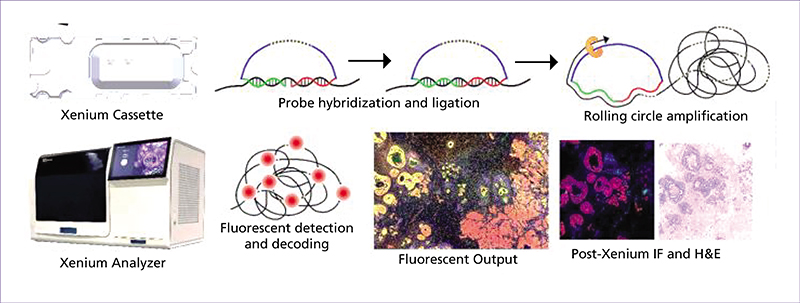
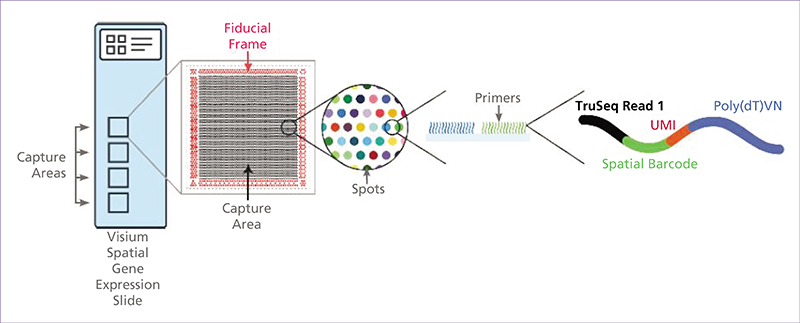
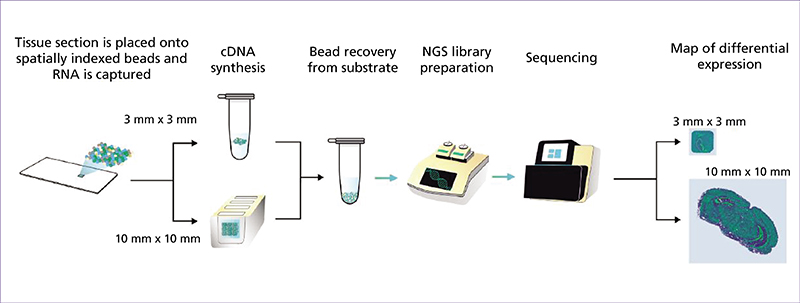
Les omiques spatiales évoluent de manière très rapide ces dernières années et, par la richesse des données fournies, elles apparaissent comme des techniques d’avenir pour comprendre les phénomènes biologiques et mieux nous soigner. Les omiques spatiales permettent d’analyser le contenu génomique, transcriptionnel, protéique ou encore métabolique à très haute résolution. Au-delà de l’identification de sous-populations moléculaires de cellules, ces approches renseignent également sur la localisation spatiale des sous-populations identifiées au sein du tissu, leur proximité entre elles et avec la matrice extracellulaire, les vaisseaux sanguins et autres composants tissulaires. La transcriptomique spatiale (TS) est un domaine de recherche émergent qui combine l’analyse génétique à haut débit avec la localisation spatiale des cellules dans les tissus. Contrairement aux méthodes classiques de séquençage, la TS offre une compréhension plus complète de l’organisation moléculaire et cellulaire des tissus. Notre revue explore différentes stratégies de profilage de TS, notamment la microdissection laser, qui permet d’isoler des cellules spécifiques d’un tissu pour des analyses omiques approfondies. Nous présentons ensuite les différentes techniques de fluorescence après hybridation in situ (FISH) utilisées en TS. Certaines techniques de dernière génération permettent également de visualiser simultanément de nombreux transcrits ciblés et des protéines avec une résolution subcellulaire 3D. Les techniques de TS reposant sur des séquençages à plus haute résolution sont aussi décrites dans un dernier chapitre. Les défis futurs pour la TS incluent l’amélioration de la résolution spatiale des méthodes basées sur le séquençage NGS (Next Generation Sequencing) et la compréhension approfondie des réseaux cellulaires et des interactions tissulaires. L’évolution rapide de ces technologies offre des opportunités passionnantes pour mieux comprendre les mécanismes biologiques complexes dans le contexte spatial des tissus.