Innovations & Thérapeutiques en Oncologie
MENUApplications cliniques du séquençage à haut débit dans les hémopathies myéloïdes Volume 3, numéro 5-6, Septembre-Décembre 2017
- Mots-clés : séquençage de nouvelle génération, mutations géniques, hémopathies myéloïdes
- DOI : 10.1684/ito.2017.0099
- Page(s) : 263-70
- Année de parution : 2017
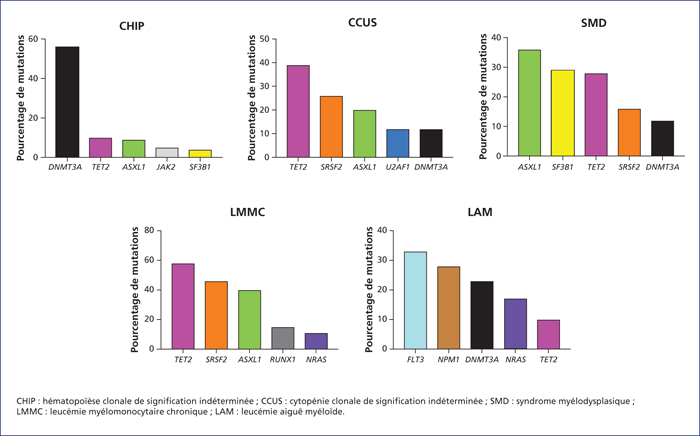
Les hémopathies myéloïdes représentent un groupe hétérogène de maladies qui partagent des bases moléculaires communes. L’utilisation du séquençage à haut débit, depuis plusieurs années, a permis d’identifier un ensemble de mutations géniques récurrentes communes aux syndromes myélodysplasiques (SMD), myéloprolifératifs (SMP) et leucémies aiguës myéloïdes (LAM). Ces mutations prennent une place croissante dans la démarche diagnostique, notamment dans le cadre de l’exploration de cytopénie(s) inexpliquée(s). Certaines mutations, comme celles de NPM1, FLT3 ou CEBPA dans les LAM, SF3B1, EZH2, RUNX1 et TP53 dans les SMD ou de ASXL1 dans les LAM, SMD, leucémies myélomonocytaires chroniques (LMMC) et myélofibroses primitives (MFP), sont associées à un pronostic particulier et sont aujourd’hui recherchées en routine. Au-delà du statut mutationnel lui-même, la proportion d’allèle muté semble influencer le phénotype et le pronostic. Enfin, ces mutations peuvent parfois guider la prise en charge thérapeutique (lorsqu’elles présentent des cibles thérapeutiques potentielles) ou bien constituer des marqueurs pour le suivi de la maladie et de son évolution.